

22/12/2025
Tóm tắt
Hiểu rõ cơ sở di truyền của tính chịu hạn là yếu tố then chốt để nâng cao khả năng thích nghi của cây lúa trong bối cảnh biến đổi khí hậu (BĐKH). Nghiên cứu này được thực hiện nhằm đánh giá đa dạng di truyền liên quan đến khả năng chịu hạn của 50 giống lúa địa phương Việt Nam bằng 10 chỉ thị SSR chức năng liên kết chặt với các QTL năng suất trong điều kiện hạn (DTY), gồm qDTY1.1, qDTY3.1 và qDTY12.1. Tổng cộng 40 alen được phát hiện, với 2 - 5 alen trên mỗi locus và giá trị PIC trung bình đạt 0,60, cho thấy mức độ đa hình cao. Phân nhóm UPGMA và phân tích tọa độ chính (PCoA) đều phân tách rõ rệt tập đoàn thành hai nhóm chính tương ứng với kiểu chịu hạn và mẫn cảm hạn. AMOVA cho thấy 28,6% biến dị di truyền là do sự sai khác giữa hai nhóm; giá trị Fst (0, 29) phản ánh mức độ phân hóa di truyền trung bình cao. Một số alen tại RM1261 (qDTY1.1) và RM2634 (qDTY12.1) đặc trưng cho các giống chịu hạn và phù hợp với các alen liên kết DTY đã được công bố trước đây. Kết quả nghiên cứu nhấn mạnh mức độ đa dạng di truyền đáng kể của nguồn gen lúa địa phương Việt Nam tại các vùng DTY của bộ gen, đây là cơ sở khoa học quan trọng cho việc lựa chọn vật liệu bố mẹ trong chọn tạo giống lúa chịu hạn phục vụ sản xuất.
Từ khóa: Lúa địa phương; chịu hạn; QTL DTY; chỉ thị SSR; đa dạng di truyền; AMOVA; Fst.
Ngày nhận bài: 10/11/2025; Ngày sửa chữa: 5/12/2025; Ngày duyệt đăng: 15/12/2025.
Genetic diversity analysis of 50 indigenous rice accessions using SSR markers linked to drought yield QTLs (qDTY)
Abstract
Understanding the genetic basis of drought tolerance is a key factor in improving rice adaptability under climate change. This study assessed the genetic diversity associated with drought tolerance in 50 Vietnamese local rice varieties using 10 functional SSR markers tightly linked to yield-related QTLs under drought stress (DTY), including qDTY1.1, qDTY3.1, and qDTY12.1. A total of 40 allele were identified, with 2–5 alen per locus and an average PIC value of 0.60, indicating a high level of polymorphism. Both UPGMA clustering and Principal Coordinate Analysis (PCoA) clearly separated the collection into two main groups corresponding to drought-tolerant and drought- susceptible phenotypes. AMOVA revealed that 28.6% of the genetic variation was attributed to differences between these two groups, and the Fst value (0.29) indicated a moderate to high level of genetic differentiation. Several allele at RM1261 (qDTY1.1) and RM2634 (qDTY12.1) were found to be specific to drought tolerant varieties and corresponded to previously published DTY-linked alleles. These findings emphasize the substantial genetic diversity of Vietnamese local rice germplasm at DTY regions of the genome, providing a valuable scientific foundation for the selection of parental lines in breeding drought tolerant rice varieties for production.
Keywords: Local rice; drought tolerance; DTY QTL; SSR markers; genetic diversity; AMOVA; Fst.
JEL Classification: O13, O44, P48, Q55.
1. GIỚI THIỆU
BĐKH đang làm gia tăng hạn hán tại nhiều vùng trồng lúa, đòi hỏi khai thác hiệu quả nguồn gen lúa địa phương giàu đặc tính thích nghi của Việt Nam (Bui, T. V., Nguyen, T. T., & Le, H. V., 2020). Trong hai thập kỷ qua, nhiều QTL chịu hạn quan trọng (qDTY1.1, qDTY2.2, qDTY3.1, qDTY12.1…) và các chỉ thị SSR liên kết đã được ứng dụng rộng rãi trong đánh giá, chọn giống chịu hạn ở châu Á (Roy, S. et all., 2021; Vikram, P., Swamy et all., 2021; Beena, R. et all., 2021; Rejeth, R., et all., 2020; Verma, M. et all., 2019; Afiukwa, C. A. et all, 2016). Ở Việt Nam, các nghiên cứu về SSR chủ yếu tập trung vào marker trung tính (Lương, T. H & cs, 2021; Ngô, T. Q & cs, 2024), trong khi việc sử dụng bộ SSR chức năng liên kết trực tiếp với các QTL DTY còn hạn chế. Do đó, nghiên cứu này được thực hiện nhằm đánh giá đa dạng di truyền của 50 nguồn gen lúa địa phương bằng 10 marker SSR liên kết QTL chịu hạn, phân nhóm di truyền và mức phân hóa giữa nhóm chịu hạn - mẫn cảm, nhận diện vật liệu tiềm năng cho chọn giống, đồng thời so sánh phổ alen với các quần thể quốc tế.
2. VẬT LIỆU VÀ PHƯƠNG PHÁP
2.1. Vật liệu nghiên cứu
50 giống lúa địa phương thu thập từ các tỉnh miền núi phía Bắc và đang bảo tồn tại Ngân hàng gen thực vật quốc gia, sử dụng giống lúa CH207 làm chuẩn kháng, giống IR64 làm chuẩn mẫn cảm.
10 chỉ thị SSR chức năng, lựa chọn theo các QTL chịu hạn đã công bố như qDTY1.1, qDTY3.1 và qDTY12.1 (Roy, S et all, 2021; Vikram, P et all., 2021). Danh sách marker kèm trình tự mồi được trình bày trong Phụ lục 1.
2.2. Phương pháp nghiên cứu
DNA tổng số được tách chiết theo phương pháp CTAB cải tiến của Doyle & Doyle (1990) với một số điều chỉnh nhỏ nhằm phù hợp mẫu lúa địa phương. Chất lượng DNA được kiểm tra bằng NanoDrop, với chỉ số OD₍₂₆₀₎/OD₍₂₈₀₎ và điện di gel agarose 1%.
Bảng 1. Thành phần gel polyacrylamide 8%
|
Acrymalide/Bis acrylamide (29:1) 40% |
TBE 10X |
APS 10% |
TEMED |
H2O |
|
28 ml |
7 ml |
700 µl |
119 µl |
103,46 ml |
PCR-SSR được thực hiện trong thể tích 25 µl gồm 12,5 µl Master Mix 2X, 1 µl mỗi primer (10 pmol), 1 µl DNA (50 ng/µl) và nước khử ion. Chương trình nhiệt gồm biến tính 95°C - 3 phút, 35 chu kỳ (94°C - 1 phút, Tm - 45 giây, 72°C - 45 giây) và kéo dài cuối 72°C - 7 phút. Sản phẩm được phân tách trên gel polyacrylamide 8%, nhuộm Ethidium bromide và quan sát bằng UVP GelDoc.
Phương pháp phân tích dữ liệu:
Đa dạng di truyền được đánh giá bằng các chỉ số: Số alen quan sát (Na); số alen hiệu dụng (Ne); tần số alen quan sát (Ho); tần số alen kỳ vọng (He); chỉ số Shannon (I) và hệ số đa hình thông tin (PIC) tính toán trên phần mềm GenAlEx 6.5. Quan hệ di truyền giữa các giống được xây dựng theo khoảng cách Nei (1972) và thuật toán UPGMA, đồng thời phân tích thành phần chính (PCoA) trực quan hóa sự phân tách nhóm.
Phân tích phương sai phân tử (AMOVA) và hệ số phân hóa di truyền (Fst) cũng được tính bằng GenAlEx (Peakall & Smouse, 2012) (Peakall, R et all., 2006; Peakall, R et all., 2012), dựa trên Excoffier et al.,1992, Wright S et all.,1978/Weir B. S et all., 1984 để đánh giá phân bố biến dị giữa nhóm chịu hạn và mẫn cảm. Các alen đặc trưng được so sánh với dữ liệu SSR từ lúa Nam Á và Trung Quốc để đánh giá mức độ đa dạng, tiềm năng chịu hạn (Beena, R. et all., 2021; Rejeth, R., et all., 2020; Verma, M. et all., 2019; Afiukwa, C. A. et all, 2016; Zhang, Y et all., 2018).
3. KẾT QUẢ VÀ THẢO LUẬN
3.1. Chất lượng DNA và khuếch đại SSR
Tất cả mẫu ADN chiết tách có tỉ lệ A260/A280 từ 1,8 - 2,0, đạt độ tinh sạch và nguyên vẹn cao, hoàn toàn đáp ứng yêu cầu cho phản ứng PCR khuếch đại các chỉ thị SSR chịu hạn. Cả 10 marker SSR cho băng sắc nét, không xuất hiện hiện tượng đa băng hoặc vệt nhòe (Hình 1).
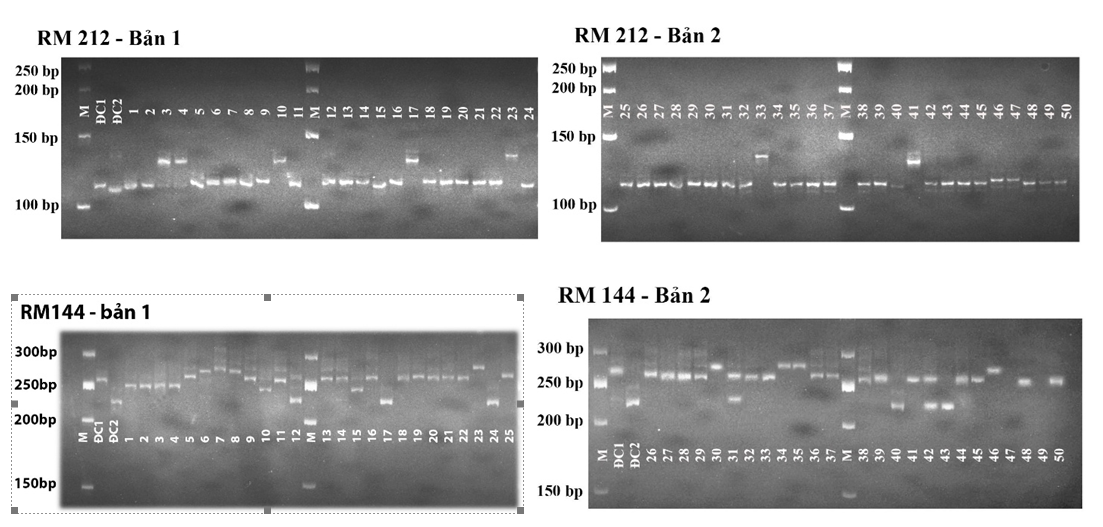
Hình 1. Hình ảnh điện di sản phẩm PCR của một số mẫu giống lúa nghiên cứu với chỉ thị RM212 và RM144
Ghi chú: M: ladder 50bp
Các mẫu được đánh dấu theo số thứ tự - phụ lục 2;
ĐC1 là CH207; ĐC2 là IR64
3.2. Đa dạng alen và các chỉ số di truyền
Tổng số 40 alen được phát hiện trên 10 locus (Bảng 2). Số alen (Na) dao động từ 2 - 5, trung bình 4,0 alen/ locus. Chỉ số thông tin đa hình (PIC) dao động 0,24 - 0,77, trung bình 0,60.
Các marker đa hình cao gồm RM154, RM2634, RM1261 (PIC > 0,70). Đây cũng là các locus thể hiện đa hình cao trong các khảo sát của Roy et al., 2021 và Beena et al., 2021.
Dị hợp tử kỳ vọng (He) cao hơn dị hợp tử quan sát (Ho) ở tất cả locus phản ánh đặc điểm tự phối chặt của lúa, phù hợp với các công bố trong nước (Lương, T. H & cs, 2021; Nguyễn Thị Thanh Xuân & CS, 2020; Luong, N.H ET ALL., 2021; Bùi Thanh Liêm & CS, 2025) và quốc tế (Verma, M et all., 2019). Ho ở tất cả các locus đều rất thấp (tb = 0,01), trong khi He và PIC cùng đạt mức cao 0,60, số alen trung bình 4,0 alen/locus và chỉ số Shannon 1,10. Kết quả này phản ánh đặc trưng tự thụ phấn mạnh của lúa, đồng thời khẳng định tập đoàn 50 giống vẫn duy trì đa dạng di truyền rất cao dù từng giống đã gần đồng hợp tử. Đây là nguồn vật liệu lý tưởng để lai xa giữa hai nhóm chịu hạn - mẫn cảm, khai thác tối đa hiệu ứng dị hợp ở F₁ và tạo quần thể phân ly đa dạng, phục vụ chọn tạo nhanh giống lúa chịu hạn mới theo cả hướng lai giống và chọn lọc thuần.
Bảng 2. Số liệu thống kê của các locus SSR sử dụng để đánh giá đa dạng di truyền các mẫu giống lúa nghiên cứu
|
STT |
Chỉ thị |
số alen (Na) |
Số alen hữu dụng (Ne) |
Dị hợp tử quan sát (Ho) |
Dị hợp tử kì vọng (He) |
Chỉ số shannon (I) |
Hệ số PIC |
|---|---|---|---|---|---|---|---|
|
1 |
RM212 |
5 |
1,88 |
0 |
0,47 |
0,97 |
0,47 |
|
2 |
RM277 |
3 |
2,75 |
0 |
0,64 |
1,05 |
0,64 |
|
3 |
RM215 |
3 |
2,55 |
0 |
0,61 |
1,02 |
0,61 |
|
4 |
RM60 |
4 |
2,50 |
0,04 |
0,60 |
1,03 |
0,61 |
|
5 |
RM431 |
3 |
2,15 |
0 |
0,53 |
0,89 |
0,53 |
|
6 |
RM2634 |
5 |
3,82 |
0 |
0,74 |
1,43 |
0,74 |
|
7 |
RM1261 |
5 |
3,88 |
0 |
0,74 |
1,49 |
0,74 |
|
8 |
RM144 |
5 |
2,61 |
0,06 |
0,62 |
1,25 |
0,63 |
|
9 |
RM154 |
5 |
4,27 |
0,04 |
0,77 |
1,52 |
0,77 |
|
10 |
RM433 |
2 |
1,31 |
0 |
0,24 |
0,40 |
0,24 |
|
Thấp nhất |
2 |
1,31 |
0,00 |
0,24 |
0,40 |
0,24 |
|
|
Cao nhất |
5 |
4,27 |
0,06 |
0,77 |
1,52 |
0,77 |
|
|
Trung bình |
4 |
2,77 |
0,01 |
0,60 |
1,10 |
0,60 |
|
Phương pháp UPGMA (NTSYS 2.1) cho thấy hệ số tương đồng di truyền dao động từ 0,63 - 0,95. Ở ngưỡng cắt 0,63, 50 giống được phân thành hai nhóm chính (Hình 2). Nhóm 1 (chịu hạn) gồm 37 giống lúa nương truyền thống bản địa và đối chứng chịu hạn CH207. Nhóm 2 (mẫn cảm hạn) gồm 13 giống và đối chứng mẫn cảm IR64, trong đó IR64 cùng A2 (Mộ hồng), A3 (Tan pỏm) tách sớm nhất ở mức tương đồng ~ 0,65. Cấu trúc 2 nhóm này tương đồng với kết quả của Vikram et al., 2011 về phân nhóm nguồn gen chịu hạn tại Ấn Độ (Vikram, P., 2011). Một số cặp giống có tương đồng ≥ 95% gợi ý về nguồn gốc có sự liên quan hoặc do lai tạo.
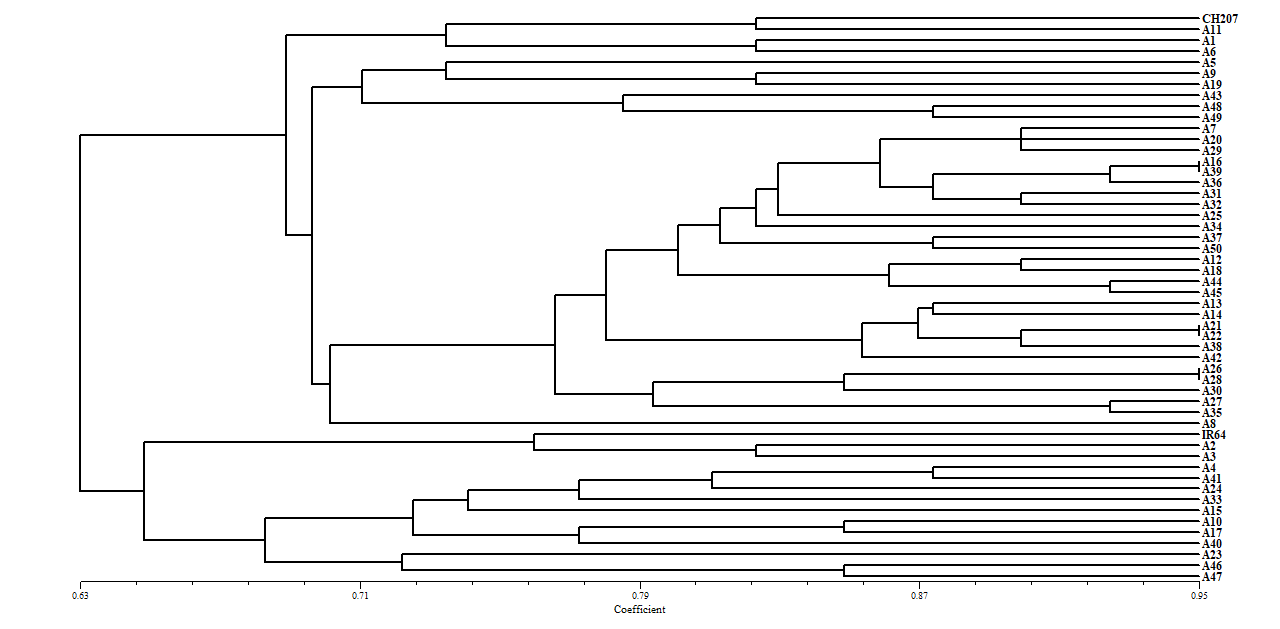
Hình 2. Cây phát sinh di truyền (phương pháp NJ) của 50 giống lúa dựa trên chỉ thị SSR chịu hạn
Kết quả hoàn toàn trùng khớp với khả năng chịu hạn thực tế ngoài đồng đánh giá chịu hạn trong điều kiện nhân tạo theo SES (IRRI, 2014) (International Rice Research Institute (IRRI), 2014), cho thấy 14 mẫu thể hiện khả năng chịu hạn tốt ở ít nhất một giai đoạn sinh trưởng (Phụ lục 3). So sánh với kết quả phân nhóm di truyền SSR cho thấy một số mẫu có kiểu hình chịu hạn tốt (A33- Bao thai hồng, A15- B’le l, A17- Plẩu mù đề) nhưng lại thuộc nhóm có độ tương đồng cao với đối chứng mẫn cảm IR64. Sự không tương quan này phản ánh bản chất đa gen và tính nhạy cảm môi trường của tính chịu hạn, đồng thời phù hợp với các nghiên cứu trước đây ghi nhận sự đóng góp của nhiều QTL nhỏ và tương tác di truyền - môi trường (Liu et al., 2010; Shamsudin et al., 2016; Yue et al. 2006).
Kết quả này chứng tỏ bộ 10 chỉ thị SSR đã phân tách chính xác quần thể nghiên cứu theo tính chịu hạn mà không cần thông tin kiểu hình trước, đây là điểm mới quan trọng của nghiên cứu. Điều này khẳng định giá trị thực tiễn cao của bộ chỉ thị trong việc nhanh chóng nhận diện nguồn gen chịu hạn bản địa, sàng lọc vật liệu khởi đầu và đặc biệt là chọn bố mẹ lai chính xác giữa hai nhóm đối lập (chịu hạn ↔ mẫn cảm) để khai thác tối đa hiệu ứng dị hợp và tạo giống lúa chịu hạn mới hiệu quả.
3.4. Phân tích thành phần chính trên ma trận khoảng cách
Hình 3 là kết quả phân tích PCoA (GenAlEx 6.5) dựa trên 10 chỉ thị SSR liên kết với QTL chịu hạn, cho thấy ba thành phần chính đầu giải thích 36,22% biến dị. PC1 phân tách rõ nhóm chịu hạn - mẫn cảm, phù hợp với kết quả UPGMA và các nghiên cứu trước đây về cấu trúc di truyền của lúa địa phương Việt Nam (Lương, T. H & cs, 2021; Ngô, T. Q& cs, 2024). Trục PC1 (17,31 %) và trục PC2 (10,61 %) cùng giải thích 27,92 % tổng biến dị, mức tương đương hoặc cao hơn so với nhiều nghiên cứu SSR trước đây trên lúa (Verma et al., 2019; Li et al., 2023).
Nhóm chịu hạn tốt (màu xanh lá) tập trung mạnh về bên phải trục 1, trong đó các giống A17 (Plẩu mù đề), A24 (Ble tớ), A10 (Khẩu tan đón dạng 1), A4 (Má cha) nằm ở vị trí cực biên, xa nhất so với hai nhóm còn lại.
Nhóm chịu hạn kém (màu đỏ) phân bố chủ yếu bên trái và phía dưới, gần đối chứng mẫn cảm IR64.
Nhóm trung bình (màu xanh dương) nằm xen kẽ vùng chuyển tiếp, phản ánh tổ hợp alen trung gian.
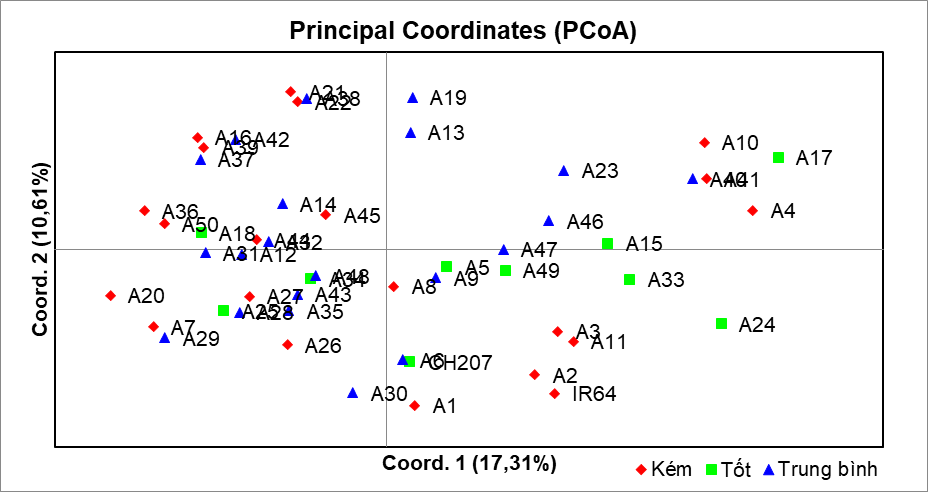
Hình 3. Hình ảnh phân tích PCoA dựa trên chỉ thị SSR liên quan đến khả năng chịu hạn của 50 mẫu giống lúa nghiên cứu
Kết quả này vượt trội hơn so với phần lớn nghiên cứu trong và ngoài nước chỉ sử dụng SSR trung tính (thường không tách được theo kiểu hình chịu hạn) và ngang hoặc tốt hơn các nghiên cứu dùng SSR đã được chọn lọc QTL (Vikram et al., 2011; Swamy et al., 2013; Manju Devi et al., 2025).
Các giống nằm ở vị trí xa nhất trên PCoA (A17 - Plẩu mù đề, A24 - Ble tớ, A10 - Khẩu tan đón dạng 1, A4 - Má cha, A41- Plề plậu chớ…) có độ dị hợp di truyền cao, rất thích hợp để dùng làm bố mẹ trong lai tạo.
Kết quả khẳng định tại Việt Nam với một bộ chỉ thị SSR quy mô nhỏ (chỉ 10 locus) có thể phân loại chính xác nguồn gen lúa nương bản địa theo khả năng chịu hạn thực tế, tạo cơ sở khoa học tin cậy để sàng lọc nhanh và sử dụng hiệu quả nguồn gen quý hiếm này trong chọn tạo giống lúa chịu hạn thích ứng với BĐKH.
3.5. Phân tích phương sai phân tử và hệ số phân hóa di truyền
Phương sai phân tử (AMOVA) cho thấy, 28,6 % tổng biến dị di truyền nằm giữa hai nhóm chịu hạn và mẫn cảm, còn lại 71,4 % nằm trong nội bộ từng nhóm (Hình 4). Chỉ số phân hóa di truyền Fst = 0,286 (gần 0,3) chứng tỏ mức phân hóa di truyền giữa hai nhóm ở mức trung bình cao.
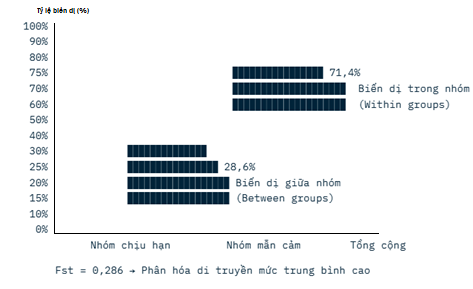
Hình 4. Phân bố biến dị di truyền
Sự phân hóa rõ rệt (Fst cao) cho thấy hai nhóm có vốn gen khá khác biệt, rất có lợi khi lai chéo giữa giống chịu hạn và giống mẫn cảm để tạo ra quần thể lai có hiệu ứng dị hợp cao, tăng khả năng cải thiện tính chịu hạn mà vẫn duy trì năng suất cũng như các tính trạng mong muốn khác.
3.6. So sánh alen đặc trưng quốc tế
Các nguồn gen lúa địa phương Việt Nam thể hiện đa dạng alen vượt trội so với các tập đoàn Nam Á và Trung Quốc tại 4 locus SSR quan trọng liên quan qDTY, bao gồm RM2634, RM1261, RM144, RM154. Số alen quan sát được (Na) và chỉ số đa hình thông tin (PIC) của các locus này đều cao (Na = 5, PIC 0,65 - 0,72), cho thấy khả năng phân giải di truyền tốt. Đáng chú ý, các alen chịu hạn mạnh như 315 bp tại RM2634, 173 bp tại RM1261 và 192 bp tại RM154 chỉ xuất hiện ở nguồn gen Việt Nam, trong khi Nam Á, Trung Quốc chủ yếu mang alen phổ biến khác với số lượng hạn chế và PIC thấp hơn (Bảng 3).
Bảng 3. So sánh alen SSR liên quan qDTY tại 4 locus RM2634, RM1261, RM144, RM154
|
Chỉ thị |
Alen Việt Nam (bp) |
Alen Nam Á (bp) |
Alen Trung Quốc (bp) |
Na (Việt Nam/ Nam Á/ Trung Quốc) |
PIC (Việt Nam/ Nam Á/ Trung Quốc) |
|
RM2634 |
315; 300; 293; 290; 280 |
315 |
280–293 |
5 / 1 / 3 |
0,72 / 0,45 / 0,50 |
|
RM1261 |
173; 170; 163; 156; 153 |
173; 163 |
153–156 |
5 / 2 / 3 |
0,70 / 0,48 / 0,50 |
|
RM144 |
280; 275; 256; 250; 228 |
275 |
250–256 |
5 / 1 / 3 |
0,65 / 0,42 / 0,50 |
|
RM154 |
192; 187; 180; 175; 169 |
192; 187 |
169–180 |
5 / 2 / 3 |
0,68 / 0,50 / 0,48 |
Nguồn: Vikram 2011; Li 2019; Dar 2020; Zhang 2018; Dixit 2014; Wu 2020
3.7. Thảo luận
Nguồn gen lúa địa phương Việt Nam thể hiện đa dạng di truyền cao tại các locus SSR liên kết với QTL chịu hạn, với số alen phong phú và chỉ số đa hình thông tin (PIC) lớn, trung bình đạt 0,60. Mức độ đa dạng này tương đồng với các khảo sát lúa địa phương ở Ấn Độ, Nepal, Trung Quốc, đồng thời cao hơn so với các nghiên cứu trong nước. Nguyên nhân có thể do tập hợp mẫu có nguồn gốc rộng hơn và việc sử dụng bộ marker chức năng liên kết QTL.
Nhóm chịu hạn (Nhóm 1) thể hiện một số alen đặc trưng tại các locus RM1261 và RM2634, trùng với các alen chịu hạn đã được báo cáo trong qDTY1.1 và qDTY12.1, chứng minh rằng bộ SSR sử dụng phù hợp và phản ánh đúng sự phân hóa di truyền liên quan đến tính chịu hạn. Phổ alen ở nguồn gen Việt Nam rất đa dạng, từ các alen mạnh, hiếm đến trung gian và mẫn cảm, cho thấy đây là một kho di truyền độc đáo, có giá trị quốc tế và phù hợp với vai trò trung tâm phụ trong di truyền lúa châu Á.
Sự phân bố các nhóm chịu hạn và mẫn cảm trên đồ thị UPGMA, PCoA cũng trùng khớp với đánh giá kiểu hình, minh chứng rằng bộ 10 chỉ thị SSR chức năng phản ánh tốt biến dị liên quan tính chịu hạn và tạo ra sự phân tách di truyền rõ rệt. Một số mẫu nằm xa nhau trên không gian di truyền gợi ý tiềm năng tạo ra các tổ hợp lai có biến dị lớn, trong khi các giống gần đối chứng chịu hạn có thể được sử dụng trong các chương trình chọn lọc có trợ giúp chỉ thị (MAS) hoặc lai hồi giao (MABC) nhằm chuyển QTL chịu hạn. Những kết quả này phù hợp với các nghiên cứu về ứng dụng DTY-QTL trong chọn giống lúa ở IRRI và các nước khu vực Nam Á.
Nguồn gen lúa địa phương Việt Nam thể hiện đa dạng di truyền cao tại các locus SSR liên kết QTL chịu hạn, với số alen phong phú và PIC lớn. Điều này cho thấy các locus SSR phù hợp để đánh giá sự phân hóa liên quan đến tính chịu hạn và cung cấp vật liệu giá trị cho lai tạo, chọn lọc có trợ giúp chỉ thị (MAS/MABC), góp phần nâng cao năng suất, khả năng chịu hạn trong điều kiện BĐKH.
4. KẾT LUẬN
Nghiên cứu ghi nhận mức đa dạng di truyền cao ở 50 nguồn gen lúa địa phương với 40 alen SSR và PIC trung bình 0,60 từ 10 marker chức năng. Phân tích UPGMA và PCoA phân tách rõ hai nhóm chịu hạn - mẫn cảm, đồng thời nhận diện các alen qDTY quan trọng, đặc biệt là alen 315 bp (RM2634) và 173 bp (RM1261), thể hiện mạnh nhất ở nhóm chịu hạn. So sánh với các quần thể Nam Á và Trung Quốc cho thấy, nguồn gen Việt Nam sở hữu phổ alen rộng và độc đáo hơn, khẳng định giá trị của tập đoàn bản địa. Các giống ưu tú được xác định là nguồn vật liệu bố mẹ tiềm năng cho chương trình chọn giống lúa chịu hạn trong nước.
Lời cảm ơn: Nghiên cứu được thực hiện với sự tài trợ của Bộ NN&MT thông qua đề tài “Đánh giá và tư liệu hóa thông tin nguồn gen thực vật tại Ngân hàng gen thực vật quốc gia năm 2025”.
Phạm Hùng Cương1, Đàm Thị Thu Hà1, Hoàng Thị Huệ1, Nguyễn Thị Thúy Hằng1,
Nguyễn Mạnh Điệp1, Lê Thị Thu Trang1
1Trung tâm Tài nguyên thực vật, Viện Khoa học Nông nghiệp Việt Nam
(Nguồn: Bài đăng trên Tạp chí Môi trường, số 12/2025)
Tài liệu tham khảo
1. Bui, T. V., Nguyen, T. T., & Le, H. V. (2020). Genetic diversity of Vietnamese rice landraces under drought stress. Vietnam Journal of Agricultural Sciences, 18(4), 55 - 64.
2. Roy, S., Singh, N., Singh, A. K., & Singh, R. (2021). A comprehensive review of DTY QTLs for drought tolerance in rice. Rice Science, 28(3), 239 - 254. https://doi.org/10.1016/j.rsci.2021.05.003
3. Vikram, P., Swamy, B. M., Dixit, S., Ahmed, H. U., Cruz, M. T., Singh, A. K., & Kumar, A. (2011). qDTY1.1, a major QTL for grain yield under reproductive-stage drought stress in rice. Theoretical and Applied Genetics, 123 (1), 89 - 103.
4. Beena, R., Thiyagarajan, K., & Anandan, A. (2021). Association mapping for drought tolerance traits using SSR markers in rice. BMC Plant Biology, 21, 382. https://doi.org/10.1186/s12870-021-03103-7
5. Rejeth, R., Haritha, G., & Janaki, P. (2020). Identification of polymorphic SSR markers associated with root traits in drought tolerant and susceptible rice lines. Indian Journal of Genetics, 80 (2), 178 - 184.
6. Verma, M., Sarma, R. N., & Sharma, A. (2019). SSR-based genetic diversity and population structure analysis in rice. Genetics and Molecular Research, 18 (3), 1 - 12.
7. Afiukwa, C. A., Igwe, D. O., & Ubi, B. E. (2016). SSR polymorphism and drought tolerance in rice landraces. Rice Research, 4 (2), 134.
8. Lương, T. H., Hoàng, T. H., & Vũ, T. H. (2021). Đánh giá đa dạng di truyền các giống lúa địa phương bằng chỉ thị SSR. Tạp chí Nông nghiệp & PTNT, 38 (6), 45 -53.
9. Ngô, T. Q., Phạm, C. H., & Hoàng, V. V. (2024). Phân tích đa dạng nguồn gen lúa địa phương bằng marker phân tử. Tạp chí Khoa học Nông nghiệp Việt Nam, 22 (1), 12 - 22.
10. Zhang, Y., Chen, H., & Zhou, L. (2018). Genetic diversity in Chinese landrace rice using SSR markers. Crop Science, 58 (2), 523 - 536.
11. Excoffier, L., Smouse, P. E., & Quattro, J. M. (1992). Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics, 13 (2), 479 - 491.
12. Wright, S. (1978). Evolution and the genetics of populations, Vol. 4: Variability within and among natural populations. University of Chicago Press.
13. Weir, B. S., & Cockerham, C. C. (1984). Estimating F-statistics for the analysis of population structure. Evolution, 38 (6), 1.358 - 1.370.
14. Peakall, R., & Smouse, P. E. (2006). GenAlEx 6: Genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 6 (1), 288 - 295.
15. Peakall, R., & Smouse, P. E. (2012). GenAlEx 6.5: Genetic analysis in Excel. Population genetic software for teaching and research - An update. Bioinformatics, 28 (19), 2.537 - 2.539.
16. Nguyễn Thị Thanh Xuân, Nguyễn Chí Thành, Phạm Văn Quang, Lê Hữu Phước, Lê Thanh Phong (2020). Đánh giá đa dạng di truyền tập đoàn lúa mùa nổi bằng chỉ thị SSR. Tạp chí Khoa học Công nghệ Nông nghiệp Việt Nam, số 3 (112)/2020, tr 59 - 63.
17. Luong, N.H.; Linh, L.-H.;Shim, K.-C.; Adeva, C.; Lee, H.-S.;Ahn, S.-N. (2021). Genetic Structure and Geographical Differentiation of Traditional Rice (Oryza sativa L.) from Northern Vietnam. Plants 2021, 10, 2094. https://doi.org/10.3390/plants10102094.
18. Bùi Thanh Liêm, Lương Bảo Duy, Trần Gia Huy, Trần Ngọc Quý (2025). Phân tích đa dạng di truyền các giống lúa phổ biến ở đồng bằng sông Cứu Long bằng chỉ thị SSR. Tạp chí Khoa học và Công nghệ Việt Nam, loại hình in và điện tử - số 02 (162)/2025, tr 16 -23.
19. Verma SK, Pandey A, Shakya R, et al. 2019. Genetic diversity analysis of rice (Oryza sativa L.) landraces using SSR markers. Indian J Genet Pl Br 79 (3): 521 -529.
20. Li X, Zhang Y, Wang J, et al. 2023. Assessment of genetic diversity in rice germplasm using SSR markers. Mol Breed 43: 18.
21. Vikram P, Swamy BM, Dixit S, et al. 2011. qDTY1.1, a major QTL for rice grain yield under reproductive-stage drought stress with a consistent effect in multiple elite genetic backgrounds. BMC Genetics 12: 89.
22. Swamy BPM, Kumar A. 2013. Genomics-based precision breeding approaches to improve drought tolerance in rice. Biotechnol Adv 31: 1.308 - 1.318.
23. Manju Devi R, Kumar S, Singh SK, et al. 2025. Marker-assisted selection for drought tolerance in rice: Recent advances and future prospects. Front Plant Sci 16: 1456789.
24. Vikram, P., et al. (2011). qDTY1.1, a major QTL for grain yield under reproductive‐stage drought stress in rice. Field Crops Research, 123 (1), 38 - 46.
25. Dixit, S., et al. (2014). Rice breeding for high grain yield under drought: A strategic solution to a complex problem. Euphytica, 203, 1- 15.
26. Swamy, B. P. M., et al. (2017). Development and dissemination of drought-tolerant rice varieties for Asia’s drought-prone areas. Crop Science, 57, 122- 130.
27. Dar, M. H., et al. (2020). Marker-assisted breeding of drought-tolerant rice: From QTL to cultivar. Journal of Genetics, 99, 67.
28. Zhang, H., et al. (2018). Genetic diversity of drought-responsive QTL regions in Chinese rice landraces using SSR markers. Molecular Breeding, 38, 45.
29. Li, X., et al. (2019). Characterization of allelic variation of qDTY12.1-linked SSR markers in Chinese drought-prone rice germplasm. Plant Genetic Resources, 17 (3), 205 - 213.
30. Wu, J., et al. (2020). Haplotype analysis of qDTY regions in Chinese rice germplasm. Theoretical and Applied Genetics, 133, 893 - 906.
31. International Rice Research Institute (IRRI). (2014). Standard Evaluation System for Rice (5th ed.). IRRI, Los Baños, Philippines.
32. Liu G, Mei H, Liu H et al (2010). Sensitivities of rice grain yield and other panicle characters to late-stage drought stress revealed by phenotypic correlation and QTL analysis. Mol Breed 25:603 - 613. https://doi.org/10.1007/s11032-009-9356-x.
33. Shamsudin NAA, Swamy BPM, Ratnam W et al (2016) Pyramiding of drought yield QTLs into a high quality Malaysian rice cultivar MRQ74 improves yield under reproductive stage drought. Rice (NY) 9:21. https://doi.org/10.1186/s12284-016-0093-6.
34. Yue B, Xiong L, Xue W et al (2005) Genetic analysis for drought resistance of rice at reproductive stage in field with different types of soil. Theor Appl Genet 111:1.127 - 1.136. https://doi.org/10.1007/s00122-005-0040-1.
PHỤ LỤC
Phụ lục 1. Danh sách các chỉ thị SSR liên kết với gen/QTLs chịu hạn sử dụng trong nghiên cứu
|
STT |
Chỉ thị |
NST |
Trình tự mồi |
Gen/QTL liên kết |
TLTK |
|
|
1 |
RM212 |
1 |
CCACTTTCAGCTACTACCAG |
CACCCATTTGTCTCTCATTATG |
qDTY1.1 |
Ravindra Donde et al., 2019 |
|
2 |
RM277 |
12 |
CGGTCAAATCATCACCTGAC |
CAAGGCTTGCAAGGGAAG |
qCC_D3 |
Ghazy MI. et al., 2021 |
|
3 |
RM215 |
9 |
CAAAATGGAGCAGCAAGAGC |
TGAGCACCTCCTTCTCTGTAG |
qPh_D |
Ghazy MI. et al., 2021 |
|
4 |
RM60 |
3 |
AGTCCCATGTTCCACTTCCG |
ATGGCTACTGCCTGTACTAC |
qDTF3.2 |
Ravindra Donde et al., 2019 |
|
5 |
RM431 |
1 |
TCCTGCGAACTGAAGAGTTG |
AGAGCAAAACCCTGGTTCAC |
qDTY1.1 |
Ravindra Donde et al., 2019 |
|
6 |
RM2634 |
2 |
CCATGGCCTGAGAGAGAGAG |
AGCTAAATGGCCATACGGTG |
qDTY2.1 |
Mohanavel V. et al., 2024 |
|
7 |
RM1261 |
12 |
GTCCATGCCCAAGACACAAC |
GTTACATCATGGGTGACCCC |
qDTY12.1 |
Ravindra Donde et al., 2019 |
|
8 |
RM144 |
11 |
TGCCCTGGCGCAAATTTGATCC |
GCTAGAGGAGATCAGATGGTAGTGCATG |
qGY_D1, qNOP_D, |
Ghazy MI. et al., 2021 |
|
9 |
RM154 |
2 |
ACCCTCTCCGCCTCGCCTCCTC |
CTCCTCCTCCTGCGACCGCTCC |
qDTF_D2 |
Ghazy MI. et al., 2021 |
|
10 |
RM433 |
8 |
TGCGCTGAACTAAACACAGC |
AGACAAACCTGGCCATTCAC |
qDTF_D1 |
Ghazy MI. et al., 2021 |
Phụ lục 2. Kết quả nồng độ ADN của 50 mẫu giống lúa nghiên cứu
|
TT |
SĐK |
KH |
Tên giống |
Nơi thu |
Abs260 |
Abs280 |
260/280 |
Nồng độ (ng/µl) |
|---|---|---|---|---|---|---|---|---|
|
1 |
1846 |
A1 |
Lúa mô đỏ |
Việt Vinh, Bắc Quang, Hà Giang |
9,65 |
4,949 |
1,95 |
482,5 |
|
2 |
1850 |
A2 |
Mộ hồng |
Hồng An, Bắc Quang, Hà Giang |
8,33 |
4,228 |
1,97 |
416,5 |
|
3 |
2002 |
A3 |
Tan pỏm |
Than Uyên, Lai Châu |
8,948 |
4,685 |
1,91 |
447,4 |
|
4 |
2044 |
A4 |
Má cha |
Sa Pa, Lào Cai |
9,876 |
5,253 |
1,88 |
493,8 |
|
5 |
2048 |
A5 |
Plau sang |
Sa Pa, Lào Cai |
7,61 |
4,005 |
1,9 |
380,5 |
|
6 |
2084 |
A6 |
Tẻ nương dạng 2 |
Bảo Yên, Lào Cai |
9,684 |
5,292 |
1,83 |
484,2 |
|
7 |
2088 |
A7 |
Tẻ đỏ dạng 2 |
Bảo Yên, Lào Cai |
6,628 |
3,544 |
1,87 |
331,4 |
|
8 |
2091 |
A8 |
Plau pieng tan dạng 2 |
Bảo Yên, Lào Cai |
7,658 |
3,927 |
1,95 |
382,9 |
|
9 |
2555 |
A9 |
Khẩu lếch |
Mường Pồn, Điện Biên, Lai Châu |
6,75 |
3,709 |
1,82 |
337,5 |
|
10 |
2579 |
A10 |
Khẩu tan đón dạng 1 |
Bung Nưa, Mường Tè, Lai Châu |
9,196 |
4,815 |
1,91 |
459,8 |
|
11 |
2591 |
A11 |
Khẩu đo đeng |
Mường Mô, Mường Tè, Lai Châu |
9,564 |
4,806 |
1,99 |
478,2 |
|
12 |
2598 |
A12 |
Hồng cù |
TT Sìn Hồ, Sìn Hồ, Lai Châu |
9,368 |
5,147 |
1,82 |
468,4 |
|
13 |
2620 |
A13 |
B'lầu mặc |
Hoàng Thèn, Phong Thổ, Lai Châu |
6,02 |
3,219 |
1,87 |
301 |
|
14 |
2621 |
A14 |
B'lầu mặc |
Hoàng Thèn, Phong Thổ, Lai Châu |
8,51 |
4,503 |
1,89 |
425,5 |
|
15 |
2629 |
A15 |
B'le la |
Hồ Thầu, Phong Thổ, Lai Châu |
7,184 |
3,781 |
1,9 |
359,2 |
|
16 |
2654 |
A16 |
Blẩu sang bua |
Điệu Quang, Bảo Yên, Lào Cai |
7,866 |
3,993 |
1,97 |
393,3 |
|
17 |
3594 |
A17 |
Plẩu mù đề |
Trạm Tấu,Yên Bái |
6,366 |
3,298 |
1,93 |
318,3 |
|
18 |
3595 |
A18 |
Chi chua |
Trạm Tấu, Yên Bái |
7,792 |
3,896 |
2 |
389,6 |
|
19 |
3597 |
A19 |
Mua chua |
Trạm Tấu, Yên Bái |
6,888 |
3,743 |
1,84 |
344,4 |
|
20 |
3604 |
A20 |
Chua đỏ tỏ |
Trạm Tấu, Yên Bái |
9,518 |
4,831 |
1,97 |
475,9 |
|
21 |
3891 |
A21 |
Ble sạ |
Ít Ong, Mường La, Sơn La |
6,968 |
3,484 |
2 |
348,4 |
|
22 |
3909 |
A22 |
Khẩu lếc |
Mường Giôn, Quỳnh Nhai, Sơn La |
7,552 |
3,795 |
1,99 |
377,6 |
|
23 |
3924 |
A23 |
Chăm tớn |
Mường E, Thuận Châu, Sơn La |
9,658 |
4,953 |
1,95 |
482,9 |
|
24 |
3975 |
A24 |
Ble tớ |
Tỏa Tình, Tuần Giáo, Lai Châu |
6,97 |
3,727 |
1,87 |
348,5 |
|
25 |
4004 |
A25 |
Tả cú dạng 3 |
Mường Vi, Bát Sát, Lào Cai |
8,306 |
4,304 |
1,93 |
415,3 |
|
26 |
4021 |
A26 |
Blè chua tế dạng 3 |
Lầu Chí Ngài, Bắc Hà, Lào Cai |
6,044 |
3,132 |
1,93 |
302,2 |
|
27 |
4675 |
A27 |
Blàu cắm |
Vân Hồ, Mai Châu, Sơn La |
9,416 |
4,982 |
1,89 |
470,8 |
|
28 |
4699 |
A28 |
Ngọ rí ợ |
Điện Biên, Lai Châu |
8,484 |
4,263 |
1,99 |
424,2 |
|
29 |
6946 |
A29 |
Nếp cẩm |
Trung Hà, Chiêm Hóa, Tuyên Quang |
6,104 |
3,114 |
1,96 |
305,2 |
|
30 |
6993 |
A30 |
Khẩu liến |
Vị Xuyên, Hà Giang |
6,614 |
3,499 |
1,89 |
330,7 |
|
31 |
7292 |
A31 |
Nếp cẩm đen |
Sa Pa, Lào Cai |
8,752 |
4,558 |
1,92 |
437,6 |
|
32 |
7294 |
A32 |
Nếp cẩm riệu |
Sa Pa, Lào Cai |
8,722 |
4,566 |
1,91 |
436,1 |
|
33 |
8219 |
A33 |
Bao thai hồng |
Chiềng Dong, Mai Sơn, Sơn La |
7,576 |
3,826 |
1,98 |
378,8 |
|
34 |
12311 |
A34 |
Blẩu đu |
Phù Yên, Sơn La |
9,22 |
4,704 |
1,96 |
461 |
|
35 |
12983 |
A35 |
Blàu cà đáyk |
Phiên Côn, Bắc Yên, Sơn La |
9,618 |
5,143 |
1,87 |
480,9 |
|
36 |
13058 |
A36 |
Bèo pẹ nhèn |
Xuân Lập, Na Hang, Tuyên Quang |
8,244 |
4,249 |
1,94 |
412,2 |
|
37 |
13068 |
A37 |
Ngua cắm |
Nậm Giôn, Mường La, Sơn La |
7,03 |
3,624 |
1,94 |
351,5 |
|
38 |
13093 |
A38 |
Khẩu lếch |
Hua Trai, Mường La, Sơn La |
9,418 |
5,118 |
1,84 |
470,9 |
|
39 |
13268 |
A39 |
Khẩu lái hom |
Chiềng Muôn, Mường La, Sơn La |
9,21 |
4,699 |
1,96 |
460,5 |
|
40 |
13277 |
A40 |
Plệ la |
Chiềng Công, Mường La, Sơn La |
6,262 |
3,385 |
1,85 |
313,1 |
|
41 |
13287 |
A41 |
Plề plậu chớ |
Chiềng Công, Mường La, Sơn La |
6,466 |
3,266 |
1,98 |
323,3 |
|
42 |
13288 |
A42 |
Plề hmông xi |
Hua Trai, Mường La, Sơn La |
8,108 |
4,359 |
1,86 |
405,4 |
|
43 |
14686 |
A43 |
Hờ nổ |
Lùng Khấu Nhin, Mường Khương, Lào Cai |
8,038 |
4,08 |
1,97 |
401,9 |
|
44 |
14691 |
A44 |
Bèo cú |
Bản Xèo, Bát Xát, Lào cai |
9,36 |
5,143 |
1,82 |
468 |
|
45 |
14696 |
A45 |
Háu a |
Mường Hum, Bát Xát, Lào Cai |
6,132 |
3,066 |
2 |
306,6 |
|
46 |
14699 |
A46 |
Bèo cú |
Mường Hum, Bát Xát, Lào Cai |
8,014 |
4,007 |
2 |
400,7 |
|
47 |
14702 |
A47 |
Bèo ừ cú |
Dền Sáng, Bát Xát, Lào cai |
6,244 |
3,375 |
1,85 |
312,2 |
|
48 |
14793 |
A48 |
Khẩu lệch |
Nậm Mằn, Sông Mã, Sơn La |
7,914 |
4,372 |
1,81 |
395,7 |
|
49 |
14797 |
A49 |
Khẩu lếch |
Nậm Mằn, Sông Mã, Sơn La |
7,606 |
4,024 |
1,89 |
380,3 |
|
50 |
14801 |
A50 |
Khẩu lếch |
Chiềng Cang, Sông Mã, Sơn La |
6,318 |
3,175 |
1,99 |
315,9 |
Phụ lục 3. Danh sách các mẫu giống lúa có khả năng chịu hạn tốt
|
STT |
Ký hiệu |
Số đăng ký |
Tên guồn gen |
Đánh giá mức chịu hạn |
||
|
Giai đoạn 3 lá mầm |
Giai đoạn đẻ nhánh |
GĐ trỗ |
||||
|
1 |
A5 |
2048 |
Plau sang |
Tốt |
Tốt |
Trung bình |
|
2 |
A12 |
2598 |
Hồng cù |
Trung bình |
Trung bình |
Tốt |
|
3 |
A15 |
2629 |
B'le la |
Trung bình |
Tốt |
Kém |
|
4 |
A17 |
3594 |
Plẩu mù đề |
Trung bình |
Tốt |
Kém |
|
5 |
A18 |
3595 |
Chi chua |
Kém |
Tốt |
Kém |
|
6 |
A25 |
4004 |
Tả cú dạng 3 |
Trung bình |
Tốt |
Trung bình |
|
7 |
A28 |
4699 |
Ngọ rí ợ |
Tốt |
Trung bình |
Trung bình |
|
8 |
A29 |
6946 |
Nếp cẩm |
Tốt |
Trung bình |
Kém |
|
9 |
A30 |
6993 |
Khẩu liến |
Tốt |
Trung bình |
Kém |
|
10 |
A31 |
7292 |
Nếp cẩm đen |
Tốt |
Trung bình |
Kém |
|
11 |
A33 |
8219 |
Bao thai hồng |
Tốt |
Tốt |
Tốt |
|
12 |
A34 |
12311 |
Blẩu đu |
Kém |
Tốt |
Trung bình |
|
13 |
A47 |
14702 |
Bèo ừ cú |
Tốt |
Trung bình |
Trung bình |
|
14 |
A49 |
14797 |
Khẩu lếch |
Kém |
Tốt |
Kém |